martes, 13 de abril de 2010
Experimento de pulso y caza
Mediante experimentos de pulso y caza, se suministran precursores radiactivos que marcan específicamente el ARN (uridina tritiada) a las células durante un breve período de tiempo (pulso). Una vez que las células han incorporado la uridina tritiada se transfieren a un medio con precursores sin marcar (caza). De este forma es posible seguir el destino del ARN marcado durante el pulso, ya que la síntesis del nuevo ARN se produce con precursores sin marcar (uridina normal). Las muestras de células tomadas después de la caza, mostraban marcaje en el núcleo, indicando que ARN se sintetiza allí, sin embargo, las muestras de células tomadas después de la caza mostraban el marcaje radiactivo en el citoplasma. Por tanto, parece que el ARN se sintetiza en el núcleo y se transporta posteriormente al citoplasma.
John Cairns y las horquillas de replicación
John Cairns (Australiano) trabajó con bacterias (E. Coli) y las cultivó en un medio con timidina titriada (H-3), luego rompió las celulas, extrajo el ADN y le hizo una autoradiografía. La autoradiografía mostró que el ADN muestra una burbuja de replicación, que crece hasta replicar totalmente el cromosoma bacteriano circular. La replicación partiria en un sitio de inicio y se expandiría en ambas direcciones a una velocidad de 45.000 nucleotidos en cada minuto a 37°C.
Meselson y Stahl y la hipótesis semi-conservativa
En 1958, Meselson y Stahl utilizaron el hecho de que cualquier soluto cuya densidad sea diferente de la de su solvente, recibe una fuerza neta en un campo gravitatorio. En un campo gravitatorio fuerte , podría haber una tendencia de los solutos, aun los pequeños, a flotar o a hundirse, según sean menos o más densos que el solvente que los rodea. Meselson y Stahl diseñaron un experimento en el cual el ADN recién sintetizado tuviera una densidad de flotación diferente de la de su progenitor (o molde) y pudiera ser así fácilmente identificable.
Se hicieron crecer cultivos de E. Coli, en un medio con nitrógeno radiactivo N15; después de lavar, se dejó que continuara el crecimiento en un medio normal con N14. El momento adecuado, se añadió el ADN aislado a una solución densa de CsCl. El tubo se colocó en una centrifuga, haciendola funcionar a velocidades suficientemente altas como para producir la sedimentación parcial del CsCl, formándose un gradiente de densidad mas denso en el fondo y menos denso arriba. En este gradiente, el ADN forma bandas en el nivel donde tiene la misma densidad que la solución salina. Después de una generación de crecimiento en el medio normal el ADN aislado forma una banda que se encuentra a mitad de camino entre el punto de igual densidad para el ADN totalmente marcado con N15 y la posición esperada para el ADN liviano normal (N14). Después de una generación de crecimiento, entonces, cada molécula de ADN tenía una mitad vieja y una mitad nueva. Calentando el ADN para separar las cadenas y luego recentrifugandolas, se hizo evidente que las moléculas hibridas tenían una cadena de polinucleotidos parental (enteramente con N15) y otra recién sintetizada (completamente con N14). En otras palabras, el ADN se replica en forma semiconservativa.
Se hicieron crecer cultivos de E. Coli, en un medio con nitrógeno radiactivo N15; después de lavar, se dejó que continuara el crecimiento en un medio normal con N14. El momento adecuado, se añadió el ADN aislado a una solución densa de CsCl. El tubo se colocó en una centrifuga, haciendola funcionar a velocidades suficientemente altas como para producir la sedimentación parcial del CsCl, formándose un gradiente de densidad mas denso en el fondo y menos denso arriba. En este gradiente, el ADN forma bandas en el nivel donde tiene la misma densidad que la solución salina. Después de una generación de crecimiento en el medio normal el ADN aislado forma una banda que se encuentra a mitad de camino entre el punto de igual densidad para el ADN totalmente marcado con N15 y la posición esperada para el ADN liviano normal (N14). Después de una generación de crecimiento, entonces, cada molécula de ADN tenía una mitad vieja y una mitad nueva. Calentando el ADN para separar las cadenas y luego recentrifugandolas, se hizo evidente que las moléculas hibridas tenían una cadena de polinucleotidos parental (enteramente con N15) y otra recién sintetizada (completamente con N14). En otras palabras, el ADN se replica en forma semiconservativa.
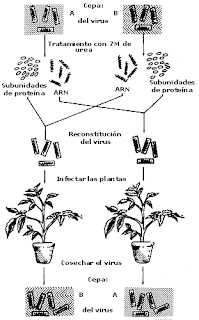
Frankel-Conrat y B. Singer
En 1957, Fraenkel-Conrat y B. Singer trabajaron con un virus vegetal (virus mosaico del tabaco, VMT). La mayoría de los virus vegetales contienen ARN en lugar de ADN. Los experimentos consistieron en unir ARN de una cepa de VMT con proteína de otra cepa de VMT, produciendo una partícula infectante hibrida. Los vegetales infectados por estas partículas produjeron virus correspondientes a la cepa que suministró el ARN al virus hibrido, demostrando que el ARN también puede transportar información hereditaria.
James Watson y Francis Crick
En 1953, los científicos James Watson y Francis Crick, analizaron los resultados obtenidos del análisis con difracción de rayos X de la molécula de ADN (obtenido por Rosalind Franklin). Esta metodología consiste en dirigir rayos X sobre fibras de ADN, de manera que al chocar con los átomos de la molécula desvían su trayectoria y son captados a través de una película fotográfica. Este tipo de análisis permitió proponer un modelo tridimensional de la molécula en forma helicoidal, el cual contiene dos cadenas paralelas y complementarias, dispuestas de manera helicoidal. Lo que no pudieron resolver en ese momento era explicar como se duplica la molécula durante la división celular.
Watson y Crick propusieron que la información genética podía conservarse durante la división celular si cada cadena de la molécula original sirviera de molde para la polimerización de una cadena complementaria en la célula hija. De esta forma, se reproduciría la secuencia de bases. Existían dos posibilidades:
Mecanismo Conservativo: La polimerización se realiza con una separación solo localizada y transitoria de las cadenas originales, dejando a la molécula progenitora intacta y originando una molécula hija completamente nueva.
Mecanismo Semi-conservativo: Si las dos cadenas del progenitor se separaran por completo, de tal manera que cada molécula hija estuviera constituida por una cadena nueva y una vieja.
Watson y Crick propusieron que la información genética podía conservarse durante la división celular si cada cadena de la molécula original sirviera de molde para la polimerización de una cadena complementaria en la célula hija. De esta forma, se reproduciría la secuencia de bases. Existían dos posibilidades:
Mecanismo Conservativo: La polimerización se realiza con una separación solo localizada y transitoria de las cadenas originales, dejando a la molécula progenitora intacta y originando una molécula hija completamente nueva.
Mecanismo Semi-conservativo: Si las dos cadenas del progenitor se separaran por completo, de tal manera que cada molécula hija estuviera constituida por una cadena nueva y una vieja.
Herchey y Chase y los virus T2
En 1952, Hershey y Chase estaban estudiando el ciclo de vida del bacteriófago T2 con la técnica de marcación isotópica. Se sabía que el virus llevaba a la célula infectada información nueva, en el sentido de que le indica a la célula que debe cesar su reproducción y , en vez de ello, debe fabricar nuevas partículas de virus. Dado que el T2 esta compuesto casi completamente de ADN y proteína, el objetivo era determinar el destino del ADN y la proteína durante la infección. Cuando se hacían crecer células infectadas por T2 en presencia de S35, se producían partículas de T2 con proteína marcada. Similarmente, cuando las células infectadas con T2 se hacían crecer en presencia de P32, se producían partículas con ADN marcado. Con el fago radiactivo, los investigadores encontraron que casi toda la proteína permanecía afuera de la célula infectada y que podía ser separada por la agitación violenta en una mezcladora domestica sin que se interrumpiera la infección. Por otra parte, se mostró que el ADN del fago entraba en la célula infectada. Dado que los genes de una partícula T2 pueden controlar la maquinaria metabólica de una célula infectada, dedicándola a la tarea de producir nuevos bacteriófagos T2, se deduce que si es el ADN del fago, y no su proteína, lo que entra al hospedador, el ADN debe llevar información genética.
Oswald Avery y la transformación con ADN
En 1944, Oswald Avery, C. MacLeod y McCarty, se propuso identificar la molécula responsable de la transformación bacteriana, descubierta por Griffith. Para esto aislaron las diferentes clases de moléculas de la cepa S (proteínas, lípidos, hidratos de carbono, ARN y ADN) y analizaron el efecto de cada una sobre el fenotipo de las células de la cepa R, al agregarlas al medio de cultivo. El resultado fue que solo una molécula, el ADN, produjo la transformación bacteriana.
Frederick Griffith y la transformación
En 1928, Friedrich Griffith utilizó dos cepas de bacterias Streptococus pneumoniae (cepa S: smooth), cuyas colonias eran de superficie encapsulada lisa y es altamente infecciosa, produciendo la muerte de ratones. Usó también una cepa R no encapsulada (rough), cuyas colonias tienen una superficie rugosa y que no produce letalidad en los ratones.
En el experimento llamado “Transformación bacteriana” demostró que las moléculas de la herencia podían pasar de una célula a otra y modificar el fenotipo de las bacterias. Griffith observó que la cepa S producía infección letal en los ratones de su laboratorio y los de la cepa R, no lo hacían. Las cepas S muertas por calor son también inofensivas, excepto cuando se las mezcla con cepa R vivas. En este último caso, se puede producir una infección fatal y en los ratones infectados se encuentran células vivas con cápsulas características de la cepa S. Este experimento permite inferir que algun factor de la cepa S muerta pasa a las cepas R vivas y las transforma en cepas infecciosas letales. Griffith no supo cual era ese factor.
En el experimento llamado “Transformación bacteriana” demostró que las moléculas de la herencia podían pasar de una célula a otra y modificar el fenotipo de las bacterias. Griffith observó que la cepa S producía infección letal en los ratones de su laboratorio y los de la cepa R, no lo hacían. Las cepas S muertas por calor son también inofensivas, excepto cuando se las mezcla con cepa R vivas. En este último caso, se puede producir una infección fatal y en los ratones infectados se encuentran células vivas con cápsulas características de la cepa S. Este experimento permite inferir que algun factor de la cepa S muerta pasa a las cepas R vivas y las transforma en cepas infecciosas letales. Griffith no supo cual era ese factor.
domingo, 11 de abril de 2010
Mecanismo de Replicación del ADN
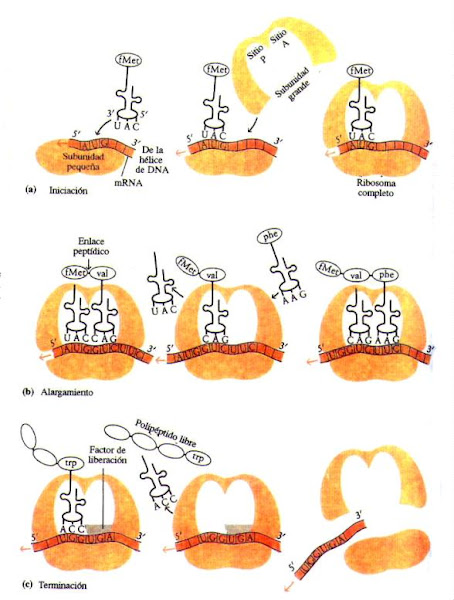
La replicación de ADN ocurre con una rapidez de 500 nucleotidos por segundo en bacterias y 50 nucleotidos por segundo en mamíferos. Estas enzimas han de ser exactas y rápidas y para ello se necesita de multiples enzimas, lo que constituye toda una maquinaria de replicación.
En 1957 se descubrió la enzima ADN polimerasa, esta enzima necesita un partidor o "primer"para polimerizar los nucleotidos en dirección 5`a 3`. La polimerización de una de las hebras ocurre de manera contínua, mientras que la hebra contraria no podía ser polimerizada con una enzima en dirección 3`-5` . Esta enzima era completamente desconocida y de hecho no existe.
Los análisis autorradiograficos hechos en los años 60 sobre cromosomas enteros en replicación marcados con timidina-H3, reveló la existencia de una región concreta de replicación que se desplaza a lo largo de la helice paterna. Debido a su estructura en forma de Y, esta región se denominó Horquilla de replicación del ADN. En la horquilla de replicación se sintetiza el ADN de las dos hebras hijas mediante un complejo multienzimatico que contiene la ADN polimerasa.
Para replicar la hebra opuesta o invertida, un japones Reiji Okazaki reveló zonas de ADN en la horquilla de crecimiento de entre 1000-2000 nucleotidos de longitud en bacterias (fragmentos de Okazaki) y más tarde se demostró su existencia en celulas eucarioticas, fragmentos de entre 100-200 nucleotidos. Estos fragmentos de Okazaki se sintetizaban unicamente en dirección 5´-3´ y que después de su sintesis se unian entre si generando largas cadenas de ADN, mediante la enzima ADN-ligasa (suelda hebras de ADN).
Una horquilla de replicación tiene una estructura asimetrica, la hebra principal o lider crece en forma contínua y la hebra opuesta o retardada crece en forma discontínua (pasos hacia atrás) con la misma ADN polimerasa III.
La fidelidad del proceso de copia durante la replicación es muy alta (1 error entre 1000 millones de pares de bases). La ADN polimerasa necesita del extremo 3´ libre de una cadena cebadora (primer) para polimerizar. Además poseen una actividad exonucleasa que elimina el apareo de un nucleotido incorrecto (autocorrección). La ADN-polimerasa requiere de un cebador en la hebra principal, por el contrario la ADN-polimerasa de la hebra opuesta completa un corto fragmento en tan solo 4 segundos, tras lo cual comienza a sintetizar otro fragmento completamente nuevo en un sitio más adelante de la cadena patrón. Para generar las cadenas primer la enzima RNA-primasa (primosoma) forma pequeños primers de 10 nucleotidos y son sintetizados a intervalos sobre la cadena retardada, para despues elongarlos mediante la ADN-polimerasa para formar el fragmento de Okazaki. La sintesis del fragmento acaba cuando choca con el otro cebador o primer. Para producir una cadena contínua a partir del gran numero de fragmento de Okazaki actua la ADN-ligasa. Los fragmentos de ARN del primer son reemplazados por ADN por la ADN-polimerasa I
Las hebras de ADN a ser replicadas son abiertas por proteínas llamadas DBP (DNA binding protein) o SSB (single strand DNA binding protein) que se unen a las hebras. La doble helice es abierta inicialmente por proteinas llamadas Helicasas (eucariontes) o DNA girasa (procariontes).
En 1957 se descubrió la enzima ADN polimerasa, esta enzima necesita un partidor o "primer"para polimerizar los nucleotidos en dirección 5`a 3`. La polimerización de una de las hebras ocurre de manera contínua, mientras que la hebra contraria no podía ser polimerizada con una enzima en dirección 3`-5` . Esta enzima era completamente desconocida y de hecho no existe.
Los análisis autorradiograficos hechos en los años 60 sobre cromosomas enteros en replicación marcados con timidina-H3, reveló la existencia de una región concreta de replicación que se desplaza a lo largo de la helice paterna. Debido a su estructura en forma de Y, esta región se denominó Horquilla de replicación del ADN. En la horquilla de replicación se sintetiza el ADN de las dos hebras hijas mediante un complejo multienzimatico que contiene la ADN polimerasa.
Para replicar la hebra opuesta o invertida, un japones Reiji Okazaki reveló zonas de ADN en la horquilla de crecimiento de entre 1000-2000 nucleotidos de longitud en bacterias (fragmentos de Okazaki) y más tarde se demostró su existencia en celulas eucarioticas, fragmentos de entre 100-200 nucleotidos. Estos fragmentos de Okazaki se sintetizaban unicamente en dirección 5´-3´ y que después de su sintesis se unian entre si generando largas cadenas de ADN, mediante la enzima ADN-ligasa (suelda hebras de ADN).
Una horquilla de replicación tiene una estructura asimetrica, la hebra principal o lider crece en forma contínua y la hebra opuesta o retardada crece en forma discontínua (pasos hacia atrás) con la misma ADN polimerasa III.
La fidelidad del proceso de copia durante la replicación es muy alta (1 error entre 1000 millones de pares de bases). La ADN polimerasa necesita del extremo 3´ libre de una cadena cebadora (primer) para polimerizar. Además poseen una actividad exonucleasa que elimina el apareo de un nucleotido incorrecto (autocorrección). La ADN-polimerasa requiere de un cebador en la hebra principal, por el contrario la ADN-polimerasa de la hebra opuesta completa un corto fragmento en tan solo 4 segundos, tras lo cual comienza a sintetizar otro fragmento completamente nuevo en un sitio más adelante de la cadena patrón. Para generar las cadenas primer la enzima RNA-primasa (primosoma) forma pequeños primers de 10 nucleotidos y son sintetizados a intervalos sobre la cadena retardada, para despues elongarlos mediante la ADN-polimerasa para formar el fragmento de Okazaki. La sintesis del fragmento acaba cuando choca con el otro cebador o primer. Para producir una cadena contínua a partir del gran numero de fragmento de Okazaki actua la ADN-ligasa. Los fragmentos de ARN del primer son reemplazados por ADN por la ADN-polimerasa I
Las hebras de ADN a ser replicadas son abiertas por proteínas llamadas DBP (DNA binding protein) o SSB (single strand DNA binding protein) que se unen a las hebras. La doble helice es abierta inicialmente por proteinas llamadas Helicasas (eucariontes) o DNA girasa (procariontes).
jueves, 6 de agosto de 2009
Enzima de restricción y ligasa
Al centro se observa el corte cohesivo de la enzima y como los ADN son asociados por la ligasa.
Abajo se observa la unión de los dos fragmentos de ADN extraños unos con otros.
Plasmidio artificial
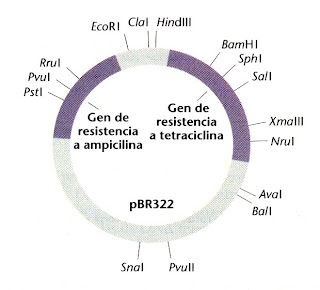
Este es un plasmidio (ADN circular) construido artificialmente y hecho a pedido para cumplir ciertas funciones que requiere el cliente. La desventaja de los plasmidios es que pueden alojar como máximo segmentos de ADN inferiores a 10.000 bases (<10kb). Particularmente en este plasmidio se introdujeron dos genes de resistencia a ampicilina y tetraciclina.
Plasmidios bacteriales
Toda bacteria posee su genoma repartido en dos tipos de cromosomas: uno es lineal, en donde están todos los genes vitales para el metabolismo de la célula; el otro es circular, llamado también plasmidio, que no posee genes esenciales para la vida de la célula, pero son útiles en casos que confieren resistencia, es decir, poseen genes cuyo producto permite sobrevivir a antibioticos del medio externo.
Cada plasmidio tiene sitios de inicio de la replicación, sitios de restricción (sitios de corte de enzimas endonucleasas), genes de resistencia a antibioticos tales como a los de Ampicilina, tetraciclina y otros.
Ingeniería Genética
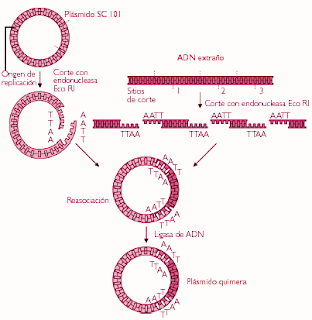
En la foto: Un plasmidio SC101 (izquierda) es cortado con una endonucleasa (enzima de restricción) al igual que el fragmento de ADN de la derecha. Los segmentos cortados son reasociados con los plasmidios y unidos con ligasas, formando el plasmidio quimera o hibrido.
La ingeniería genética consiste en la modificación del ADN de un organismo para producir nuevos genes con nuevas características. Esta tecnología ha revolucionado la medicina, la agricultura, la farmacia, la genetica y las investigaciones básicas en biológía y evolución.
ADN recombinante: Esta es una técnica para combinar segmentos de ADN de distinto origen, permitir nuevas combinaciones de genes mediante su aislamiento e inserción en otro organismo.
Las herramientas son:
1. Enzimas de restricción: Algunas enzimas en bacterias tienen la misión de atacar segmentos de ADN extraño en una bacteria como por ejemplo ADN viral. Estas enzimas hoy son utilizadas como tijeras moleculares que cortan en secuencias específicas. Estos cortes pueden ser cohesivos o romos. A estas enzimas se les conoce como enzimas de restricción (XbaI, Hpat, BamHI,SalI, EcoR1).
2. Enzimas Ligasas: Son enzimas capaces de ligar o unir dos extremos del mismo ADN o de distintos fragmentos de ADN.
3. Vectores: Un vector es un vehículo en el cual se transporta el gen de interés. El vector más común utilizado es el Plasmidio (ADN circular en bacterias). Estos plasmidios se aislan, se le incorporan genes de interes para estudiar y luego se regresan a las bacterias para que por cada replicación también replique el plasmidio con el gen de interés. De esta manera se pueden sintetizar millones de copias del gen.
4. Celulas huesped: Los huesped más comunes usados son algunas bacterias ampliamente conocidas y estudiadas. Estas bacterias sirven como fuente de plasmidios y como receptor de plamidios ajenos con inserciones de genes de interes, de origen bacterial, plantas o humano.
Regulación génica (Caso Operones)
 En la foto: Francois Jacob y Jacques Monod
En la foto: Francois Jacob y Jacques MonodNormalmente no se pierden cromosomas ni ningun gen en las celulas del cuerpo, sin embargo, claramente las celulas musculares, hepáticas, renales y cerebrales son distintas en su forma y función y fabrican diferentes productos. La razón de ello es que sólo parte de la información en los genes se expresa en una determinada célula. Algunos genes son inactivos, otros tienen gran actividad y otros se expresan en distintas ocaciones a lo largo del desarrollo. Por ejemplo en Echerichia coli existen alrededor de 4000 genes, algunos codifican proteínas necesarias para el ciclo de vida (glicolisis), mientras que otros son necesarios para vivir en condiciones especiales de crecimiento (enzima lactasa que degrada la lactosa de la leche).
Normalmente hay dos mecanismos básicos para controlar la actividad metabólica:
a) puede regular la actividad de algunas enzimas (eficiencia)
b) se puede controlar el número de moleculas enzimaticas (cantidad)
Francois Jacob y Jacques Monod en 1961 demostraron la regulación génica y propusieron la existencia del Operon como unidad de regulación de la transcripción.
Una bacteria sometida a un medio con glucosa, incorpora este nutriente para realizar la respiración celular. En ausencia de glucosa y presencia de lactosa en el medio, se incrementa la sintesis de dos enzimas, la lactasa y la beta-galactosidasa. La lactasa rompe la lactosa en glucosa y galactosa, mientras que la beta-galactosidasa transforma la galactosa en glucosa. De esta manera en presencia unicamente de lactosa, la celula se las arregla para producir glucosa para su respiración celular.
Muchos genes se caracterizan por tener una secuencia Operador (O), una secuencia promotora (P) y luego el gen. El operador puede unir una proteína represora que bloquea al promotor. El promotor permite la unión de la enzima RNA polimerasa para permitir la transcripción en el sitio de inicio.
Operon Lactosa (E. coli): La lactosa actúa como molécula inductora, es decir, un sistema apagado se enciende.
a) Ausencia de lactosa y presencia de glucosa en el medio
Sin Lactosa, una proteína Represora se une al operador libremente bloqueando al promotor e impide la unión de la RNA-polimerasa a este sitio, y por lo tanto no se transcriben los genes beta-galactosidasa, permeasa y trasacetilasa, necesarios para tratar a la lactosa.
b) Presencia de Lactosa y ausencia de glucosa en el medio
La permeasa permite el transporte de la lactosa a través de la membrana hacia el citoplasma. Una vez adentro la lactosa se une al represor para cambiar su conformación y lo suelta del sitio operador, desbloqueando así el promotor y permitiendo que la RNA polimerasa se una a este y transcriba los genes beta-galactosidasa, permeasa y transacetilasa. La beta-galactosidasa cumple la función para la cual fue sintetizada, transformar la galactosa en glucosa.
Operon Triptofano (Salmonella tiphi). El triptofano es una molecula represible, es decir, un sistea encendido lo apaga.
a) En presencia de Triptofano (co-represor), este aminoácido se une a una proteína represora inactiva y la convierte en activa. El represor y el co-represor se unen a su sitio operador y bloquean el promotor, impidiendo la unión de la RNA polimerasa e impidiendo además la transcripción de 5 genes de este Operón.
b) En ausencia de Triptofano, el represor queda inactivo y los 5 genes se transcriben normalmente por la RNA polimerasa.
miércoles, 10 de junio de 2009
Maduración del ARNm en eucariontes
 Una vez que se ha sintetizado el ARNm, en las celulas eucarioticas, esta molécula sufre una serie de modificaciones dentro del núcleo, antes de ser transportado al citoplasma. Este proceso se denomina maduración del ARNm y consiste en la eliminación de segmentos de ARNm que no participan en la síntesis de proteínas. Estos segmentos denominados INTRONES, son eliminados por enzimas especiales dentro del núcleo.
Una vez que se ha sintetizado el ARNm, en las celulas eucarioticas, esta molécula sufre una serie de modificaciones dentro del núcleo, antes de ser transportado al citoplasma. Este proceso se denomina maduración del ARNm y consiste en la eliminación de segmentos de ARNm que no participan en la síntesis de proteínas. Estos segmentos denominados INTRONES, son eliminados por enzimas especiales dentro del núcleo.Los segmentos de ARN que participan en la síntesis de proteínas se llaman EXONES, y son unidos entre sí por un conjunto de enzimas presentes también en el núcleo celular. Por lo tanto este proceso consiste en el corte de intrones y el empalme de exones, lo que determina que la molecula de ARNm recién transcrita (ARNhn), sea más larga que la molécula de ARNm maduro. El proceso de corte y empalme se desarrolla mediante un mecanismo de splicing, en presencia de un complejo formado por proteínas y ARN, llamado speisosoma.
Suscribirse a:
Entradas (Atom)